今日は主座標分析(Principal Coordinate Analysis; PCoA)の紹介を簡単にしたいと思います。
主座標分析は古典的多次元尺度構成法(Classical Multidimensional Scaling; CMDS)とも呼ばれる統計解析手法です。
この解析手法を使用する主な目的は、高次元のデータを2次元や3次元に落として視覚化したいという時に使います。
以前紹介した主成分分析と同じような感じですね。*1
主成分分析との違いを簡単に言うと、主成分分析はユークリッド距離をなるべく保ちながら低次元に落とす方法ですが、主座標分析はユークリッド距離だけでなく、他の距離や類似度*2が使えるという点にあります。
例えば、ユークリッド距離の代わりに相関係数を使えば、相関の高いもの同士が近い配置になるようなプロットを作ることが可能です。
データを用意する
さっそくやってみたいのですが、その前に今回解析するデータを紹介しておきます。
今回使うデータは、データセットまとめでも紹介した eurodist データセットです。
このデータには、ヨーロッパの21都市間の道路距離が示されています。
下に一部を示します。アテネからバルセロナへ行くには 3313 km の道路を通る必要があるみたいですね。
| アテネ | バルセロナ | ブリュッセル | カレー | シェルブール | ケルン | |
|---|---|---|---|---|---|---|
| アテネ | 0 | |||||
| バルセロナ | 3313 | 0 | ||||
| ブリュッセル | 2963 | 1318 | 0 | |||
| カレー | 3175 | 1326 | 204 | 0 | ||
| シェルブール | 3339 | 1294 | 583 | 460 | 0 | |
| ケルン | 2762 | 1498 | 206 | 409 | 785 | 0 |
このデータは高次元データではなく、単なる距離データです。
主座標分析は距離データを分析するので入力が高次元データである必要はありません。
例えば、文字列どうしを編集距離で比較したときの距離行列を入力とすることもできます。
高次元データを解析する場合は、その距離行列をあらかじめ求めておく必要があります。
やってみよう
統計言語 R では、主座標分析は cmdscale() 関数により実行することができます。
cmdscale() 関数の入力は、各サンプル間の距離を示した行列です。
eurodist データセットは dist オブジェクトなのでまずは行列に変換します。
data(eurodist) data <- as.matrix(eurodist)
このデータに対して主座標分析を行い、グラフにプロットしてみましょう。
result <- cmdscale(data, k = 2) plot(result, type = "n") text(result, labels = rownames(result))
比べてみよう
さて、この結果がうまくいったかどうかを調べるために、少し小細工をします。
citynames <- c("アテネ", "バルセロナ", "ブリュッセル", "カレー", "シェルブール", "ケルン", "コペンハーゲン", "ジュネーブ", "ジブラルタル", "ハンブルク", "オランダのフック", "リスボン","ライオンズ", "マドリード", "マルセイユ", "ミラノ", "ミュンヘン", "パリ","ローマ", "ストックホルム", "ウィーン") colors <- c(2, 4, 1, 1, 1, 1, 6, 1, 1, 6, 1, 3, 1, 3, 4, 4, 5, 5, 2, 6, 5) plot(result, type = "n") text(result, labels = citynames, col = colors, cex = 0.8, font = 2)
都市名を日本語にして、いくつか色をつけてみました。
これを Google Map の地図と比較してみます。

どうやら上下が逆のようですね。
主座標分析において軸の向きに意味はないので反転させてみましょう。
plot(result[,1], -result[,2], type = "n") text(result[,1], -result[,2], labels = citynames, col = colors, cex = 0.8, font = 2)
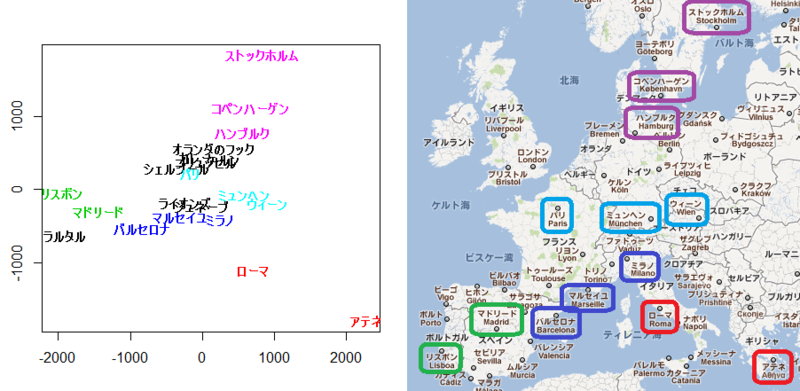
どうでしょうか?
よく一致していると思いませんか?
これはデータとして都市間の距離だけをもとにして2次元座標を復元しているのです。すごい!
縮約できる次元の限界
ところで、データによっては距離を保ったまま低次元へマッピングすることが難しい場合もあります。
これを判断する基準に Mardia の第一基準と第二基準というものがあります。
それぞれ以下のようにして求めることができます。
k <- 2 # 縮約したい次元数 result <- cmdscale(data, k = k, eig = TRUE) # 固有値を計算するように指定 eig <- result$eig # 求まった固有値 p1 <- sum(abs(eig[1:k])) / sum(abs(eig)) # Mardia 第一基準の計算 p2 <- sum(eig[1:k] ^ 2) / sum(eig ^ 2) # Mardia 第二基準の計算 cat("Mardia fit measure 1 = ", p1, "\n") cat("Mardia fit measure 2 = ", p2, "\n")
Mardia fit measure 1 = 0.7537543
Mardia fit measure 2 = 0.977388
この値のどちらかが 0.8 以上であれば、その次元に縮約したときの距離の誤差が許容範囲であると考えられます。
今の場合でいうと、eurodist データを2次元に縮約した場合、Mardia の第一基準は満たしていませんが、第二基準を満たしているので OK ということになります。
もし、どちらも満たしていない場合は、次元数を上げる必要があります。
まとめ
主座標分析について簡単に紹介してみました。
主座標分析は、サンプル間の距離行列が与えられたとき、その距離を保ったまま低次元にマッピングして視覚化する手法です。
この手法はわりと応用範囲の広い優れモノです。
特に高次元データに対して、任意の距離関数でクラスターを形成するかどうか調べるのに使えます。
私が今やっている仕事では、グループ間の比較をするためにマハラノビス汎距離で主座標分析したりしています。
もっと知りたいという方は、Rと多次元尺度法(PDF) が参考になると思います。
それでは。
*1:主成分分析が簡単にできるサイトを作った http://d.hatena.ne.jp/hoxo_m/20120106/p1 参照
*2:正確には非類似度
